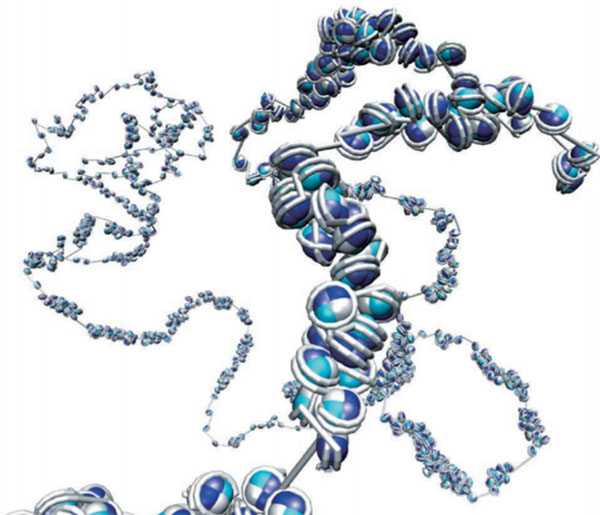
生命の遺伝情報を担うゲノムは、細胞内において、160〜200塩基対毎にヒストン8量体に巻きついて形成される“ヌクレオソーム”を最小構造単位として存在している。しかしながら、実際の細胞内でこのヌクレオソームがどのように並んで存在しているのかは、これまでの研究ではあまりよくわかっていなかった。そこで私たちは、ゲノムの3次元構造をヌクレオソーム分解能で決定する手法の開発に取り組み、これに成功した。
この開発のため、従来の次世代シーケンサーを用いた実験法(Hi-C法)の高分解能化を行うと共に、ゲノム内の全てのヌクレオソームを3次元モデリングする新たな計算法の開発を行った。実験法の開発では、ゲノム上の各ヌクレオソームのDNA巻きつき開始・終了点間の近接関係をそれぞれ解析できる方法を構築した。これに対し計算法の開発では、大規模な分子動力学計算をスーパーコンピューター上で実験データに基づいて行い、各ヌクレオソームの位置と配向を含む全ゲノムの3次元分子構造を決定する方法を構築した。開発した技術は、”Hi-C” with nucleosome “O”rientationの略からと、さらに「配向」が解析できる特徴から、「Hi-CO」法と名付けた。
結果、1つ1つのヌクレオソームのレベルから全染色体のレベルに至る、ゲノムの階層構造が初めて実験的に明らかになった(図1)。面白いことに、出芽酵母のゲノム構造の解析を行ったところ、これまで規則的に並んでいると考えられていたヌクレオソームの配列が、実は2通りのヌクレオソーム配列(正四面体型とひし形型)の組み合わせによって成り立っていることが見えてきた。タンパク質の折り畳み構造の基本構造であるαヘリックス・βシートにちなんで、両者をαテトラへドロン・βロンバス構造と命名した。
さらには、ヌクレオソームの配置構造が、遺伝子の発現制御の性質と密接に関連して有意に変化していることを見つけた。この結果は、細胞内のゲノムがどのようにして分化や発生などの際に、それぞれの遺伝子の発現をコントロールしているか、その分子機序を知るための重要な基礎となると考えられる。今後、ヒトを含む様々な生物種に解析を拡張することにより、ヌクレオソーム配置構造とゲノム機能のさらなる詳細な相関性や、ゲノム構造による遺伝子発現の制御原理、疾患や薬剤存在下におけるゲノム構造の可変性などが明らかになってくると期待される。
Hi-CO法の開発は当時理化学研究所にあった私の研究室で8年の期間を費やして行いました。開発を初めて4年経ったときに、Hi-CO法と競合する論文が他の研究室から出されてしまい、当時は大いに焦りましたが、その後の分析から実は私たちの方法のほうが高い分解能を有することを見つけ、さらには計算機シミュレーションを専門とする方と一緒にヒストンやDNAの物理的性質を加味した3次元構造決定を行うアルゴリズムを新たに開発することで、より良い形で論文を報告することができました。長期に渡る開発に付き合って頂いた第一著者の大野さんをはじめとする研究室のメンバー(図2)、並びに第二著者の安藤さんをはじめとする共同研究者の方々には本当に感謝しています。